Bakteriofag T4 to jeden z najlepiej opisanych bakteriofagów. Został on w pełni zsekwencjonowany już w latach 80 XX wieku. Jest bardzo dobrym modelem badawczym zarówno w biologii molekularnej, jak i w terapii fagowej. Jest sztandarowym przykładem faga przeprowadzającego cykl lityczny.
Spis treści
Charakterystyka bakteriofaga T4
Bakteriofag T4, jest jednym z siedmiu fagów (T1 – T7, typ T), które infekują bakterie Escherichia coli [Ryc. 1]. Fag ten należy do rodziny Straboviridae i rzędu Caudoviricetes (do 2020 roku należał do rodziny Myoviridae i rzędu Caudovirales ze względu na to, że charakteryzuje się kurczliwością ogonka – zmiany taksonomiczne wirusów można śledzić na stronie ictv.global).
Budowa bakteriofaga T4
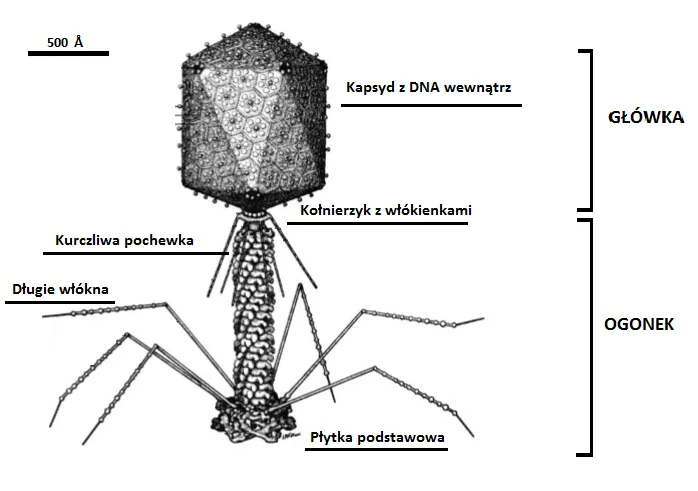
Dojrzała cząstka wirusa (wirion) zbudowana jest z wydłużonej ikosaedralnej główki i ogonka, który pokryty jest kurczliwą osłonką i zakończony płytą podstawową, do której przyłączone jest sześć włókienek [Ryc. 2]. Genom bakteriofaga T4 ma wielkość 168 kbp, zawiera 289 otwartych ramek odczytu (ORF – Open Reading Frames) i składa się z dwuniciowego DNA (dsDNA). Znajduje się on w główce bakteriofaga.
[J. A. Braga de Andrade et al., 2011]
Główka bakteriofaga T4
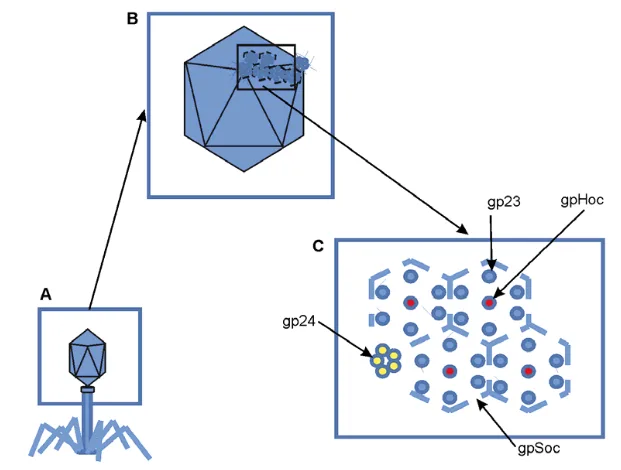
Główka faga T4 to inaczej kapsyd. Jest to białkowa osłona otaczająca materiał genetyczny bakteriofaga. Główka bakteriofaga T4 ma długość 120 nm i szerokość 86 nm. Składa się z 3 podstawowych białek:
- gp23 (gp – produkt genu) – tworzy sześciokątną sieć kapsydu;
- gp24 – tworzy pentamery na 11 z 12 wierzchołków;
- gp20 – tworzy unikalny dodekameryczny wierzchołek portalu, przez który przechodzi DNA i jest miejscem połączenia z ogonkiem.
Powierzchnię kapsydu pokrywają dwa białka:
- Hoc – wysoce antygenowe białka zewnętrzne kapsydu o masie 40 kDa, w około 160 kopiach;
- Soc – małe zewnętrzne białka kapsydu o masie 9 kDa, w około 960 kopiach.
Oba białka nie są konieczne do przeżycia faga oraz nie wpływają na przebieg infekcji komórek bakteryjnych przez faga. W różnych źródłach można znaleźć inną ilość kopii białek Hoc
i Soc.
Krótkie włókna faga
W miejscu łączenia główki i ogonka znajduje się 6 włókien zbudowanych z gpWac (białko regulujące antygen włosków), oraz kołnierzyk utworzony z białek gp13 i gp14. Włókienka odpowiadają za rozpoznawanie warunków środowiska i chronią przed zaadsorbowaniem faga do powierzchni komórki gospodarza w nieodpowiednim momencie, np. w zbyt niskiej temperaturze.
Ogonek faga T4
Ogonek faga T4 składa się z dwóch koncentrycznych cylindrów białkowych – kanału centralnego i kurczliwej pochewki. Zakończony jest płytką podstawową i włóknami. Wewnętrzny cylinder białkowy, który nazywamy kanałem centralnym, zbudowany jest ze 144 kopii białka gp19, a zakończony jest heksametrem białka gp3. Kurczliwa pochewka składa się ze 144 kopii białka gp18 i powstaje poprzez polimeryzację wokół kanału głównego. Do ostatniego rzędu białek gp3 i gp18 przyłącza się heksamer białka gp15 kończąc budowę ogonka.
Na końcu ogonka znajduje się płytka podstawowa, do której przytwierdzone są długie włókna ogonka odpowiedzialne za rozpoznawanie receptorów bakterii E. coli. Natomiast pod płytką podstawową znajdują się krótkie włókna ogonka, dzięki którym fag może zakotwiczyć się do lipopolisacharydowych (LPS) receptorów powierzchniowych obecnych na powierzchni komórki.
Długie włókna ogonka
Są sensorami środowiskowymi i odpowiadają za adsorpcję do komórki gospodarza. Połączone są z płytką podstawową za pomocą białek gp7 i gp9. Włókna te mają około 1450 Å długości oraz 40 Å średnicy. Każde włókno składa się z dwóch części: bliższej i dalszej, które są sztywne i połączone rejonem zawiasowym.
Bliższą część włókna tworzą białka gp34, natomiast dalszą tworzą białka gp35, gp36 i gp37. Białko gp12, które znajduje się w krótkich włóknach ogonka pod płytką podstawową i białko gp37 mają homologiczne C-końcowe regiony. Jest tak prawdopodobnie dlatego, że oba pełnią podobną funkcję, czyli wiążą się do LPS komórki E. coli, dzięki czemu bakteriofag może rozpoznać i zainfekować gospodarza.
Wszystkie elementy faga: główka, ogonek, krótkie włókna i długie włókna ogonka powstają oddzielnie. Dopiero po utworzeniu poszczególnych elementów następuje proces zwany montażem i w ten sposób powstają potomne wiriony.
Cykl życiowy faga T4
Bakteriofag T4 przeprowadza cykl lityczny po infekcji bakterii E. coli (jest zdolny do infekcji także innych Gram-ujemnych bakterii). Po wstrzyknięciu materiału genetycznego do wnętrza komórki i jego namnożeniu dochodzi do lizy komórki bakteryjnej.
Adsorbcja
Bakteriofag T4 rozpoznaje na powierzchni komórki E. coli LPS albo OmpC (antygen powierzchniowy). Kiedy przynajmniej 3 długie włókna zwiążą się z receptorami komórki gospodarza (adsorpcja), to zmieniają one swoją konformację. Płytka podstawowa znajduje się wtedy w bliskim sąsiedztwie z powierzchnią komórki i krótkie włókna ogona mogą oddziaływać z receptorami komórki gospodarza. Następnie konformacja płytki podstawowej zmienia się z heksagonalnej w gwieździstą. Dochodzi wtedy do kurczenia się pochewki ogonowej. Sztywna rurka ogonowa, która jest ostro zakończona, przedostaje się przez zewnętrzną błonę komórkową gospodarza. Kiedy dochodzi do zetknięcia się rurki ogonowej z peptydoglikanem uwalniany jest lizozym faga T4, który trawi warstwę peptydoglikanu. Tworzy się wtedy wejście, przez które rurka ogonowa może dostać się do cytoplazmy gospodarza i po zetknięciu z nią następuje uwolnienie fagowego DNA do gospodarza.
Replikacja i montaż faga
Na początku infekcji dochodzi do zmian w błonie komórkowej bakterii, gdzie ekspresji ulegają fagowe geny imm. Przez pierwsze 4 minuty infekcji, jeśli dojdzie do dodatkowej infekcji to DNA fagów spokrewnionych z fagiem T4 ulegnie strawieniu w peryplazmie. Dochodzi do ekspresji innych genów faga odpowiedzialnych za produkcję wszystkich komponentów wirusa, następnie dochodzi do złożenia wirionów, gdzie powstaje około 100-150 cząsteczek wirusa.
Uwolnienie wirionów potomnych
Następuje aktywacja genów kodujących białka gpe (lizozym) i gpt (holina). Holina tworzy otwory w błonie, przez które wydostaje się lizozym i tnie peptydoglikan. Potomne wiriony mogą uwolnić się poza komórkę bakteryjną. Regulacja procesu lizy została już wcześniej opisana w artykule o holinach.
Zastosowanie bakteriofaga T4
Tak jak już wcześniej opisany bakteriofag lambda fag T4 przyczynił się do rozwoju wiedzy z zakresu biologii molekularnej. Badania nad nim doprowadziły między innymi do:
- przeprowadzania miejscowo-specyficznej mutagenezy i analizy wielu ważnych genów, w tym tych, które kodują takie enzymy jak lizozym faga T4, polimerazę DNA, czy białko regA,
- rozwoju systemu phage-display (bardzo wiele białek o różnym rozmiarze i strukturze można przyłączyć do kapsydu tego faga),
- rozwoju szczepionek (np. szczepionka przeciw wirusowi vvIBDV, który powodował wirusowe zapalenie stawów u kurczaków),
- rozwoju terapii fagowej (mutacje w białkach główki, tak by fag nie był immunogenny).
Istnieje wiele możliwości rozwoju przy wykorzystaniu faga T4 jako modelu badawczego. Na zajęciach laboratoryjnych z mikrobiologii bakteriofagi Lambda oraz T4, to nadal najczęściej wykorzystywane fagi i sztandarowe przykłady wirusów bakteryjnych.
Literatura
- Jacy Alves Braga de Andrade, Edna Freymüller, Ulysses Fagundes-Neto (2011). Adherence of enteroaggregative Escherichia coli to the ileal and colonic mucosa: an in vitro study utilizing the scanning electron microscopy. Arquivos de Gastroenterologia, 48, DOI: 10.1590/S0004-28032011000300009
- Moh Lan Yap i Michael G Rossmann (2014). Structure and function of bacteriophage T4. Future Microbiology, 9, 1319–1327, DOI: 10.2217/fmb.14.91
- Mariam Gamkrelidze i Krystyna Dąbrowska (2014). T4 bacteriophage as a phage display platform. Arch Microbiol 196, 473-479, DOI: 10.1007/s00203-014-0989-8
- P. G. Leiman, S. Kanamarua,V.V. Mesyanzhinovb, F. Arisakac i M. G. Rossmanna (2003). Structure and morphogenesis of bacteriophage T4. Cellular and Molecular Life Sciences, 60, 2356-2370, DOI: 10.1007/s00018-003-3072-1
- https://ictv.global/taxonomy/taxondetails?taxnode_id=202200332
- Kurzępa, A., Dąbrowska, K., Świtała-Jeleń, K., & Górski, A. (2009). Molecular modification of T4 bacteriophage proteins and its potential application. Folia Microbiologica, 54, 5-15. DOI:10.1007/s12223-009-0002-0.
- Miller, E. S., Kutter, E., Mosig, G., Arisaka, F., Kunisawa, T., & Rüger, W. (2003). Bacteriophage T4 genome. Microbiology and molecular biology reviews, 67(1), 86-156. DOI:10.1128/MMBR.67.1.86–156.2003
