Terapia fagowa po odkryciu antybiotyków została odepchnięta na dalszy plan. Jednak badania nad fagami nie ustały i udało się wykazać, że fagi można wykorzystać na wiele innych sposobów. Przykładem jest bakteriofag lambda (λ) oficjalnie odkryty w 1951 roku przez Esther Lederberg. Badania nad fagiem λ przyniosły więcej korzyści niż ktokolwiek mógłby się spodziewać. Skąd to zainteresowanie fagiem i jego wielki fenomen? Postaram się to wszystko wyjaśnić.
Spis treści
Opis faga lambda
Bakteriofag λ został wyindukowany z Escherichia coli K12. Łysinki wytwarzane przez tego wirusa są mętne. W ikosaedralnym kapsydzie znajduje się materiał genetyczny faga, czyli dwuniciowy DNA (dsDNA) zbudowany z 48 502 par zasad (pz) z dwunastonukleotydowymi jednoniciowymi komplementarnymi końcami. Główka faga ma średnicę około 650 Å. Ogonek faga jest podłużny o szerokości 120 Å, ale w odległości 200 Å od dalszego końca zwęża się do cienkiego włókna. Ogonek jest podzielony na 35 prążków i ma całkowitą długość 1500 Å. Jest to obraz typowy dla fagów Siphoviridae, do których fag lambda został zaklasyfikowany.
W marcu 2021 roku ICTV wprowadziło kilka zmian w taksonomii wirusów. Usunięto rząd Caudovirales i rodziny Myoviridae, Podoviridae i Siphoviridae, które do tego rzędu należały. Bakteriofag lambda jako gatunek znaleźć można teraz pod nazwą Lambdavirus lambda w klasie Caudoviricetes.
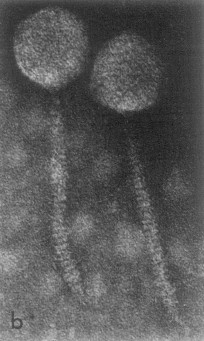
Mechanizm adsorpcji i wniknięcia do komórki bakteryjnej
Bakteriofag λ adsorbuje się do powierzchni komórki gospodarza, E. coli, po rozpoznaniu receptora Lam B. Receptor zlokalizowany jest w błonie zewnętrznej komórki bakteryjnej i odgrywa rolę w transporcie maltozy. Materiał genetyczny faga dsDNA dostaje się do cytoplazmy komórki gospodarza przez jego niekurczliwy ogonek a dalej dzięki działaniu układu enzymatycznego E. coli. Ten układ składa się z białek PtsP i PtsM znajdujących się w błonie wewnętrznej i zwykle wykorzystywanych do transportu mannozy.
Wybór cyklu życiowego
Po wniknięciu DNA faga do cytoplazmy gospodarza następuje cyrkularyzacja DNA i zapada decyzja, czy bakteriofag przeprowadzi cykl lityczny, czy lizogeniczny. Bierze w tym udział specyficzny fagowy system regulacyjny i wykorzystuje wiele czynników zależnych od faga i bakterii (np. temperatura, stan głodu komórkowego, etc.). Mówiąc bardzo krótko w przypadku rozwoju cyklu litycznego produkowane jest białko Cro a w przypadku lizogenicznego białko CI.
W przypadku lizogenizacji DNA wbudowuje się do chromosomu gospodarza i tam pozostaje w formie zwanej profagiem. Taki stan może pozostać przez wiele generacji. Cykl lityczny może się rozpocząć po indukcji profaga, np. jakimś stresorem dla komórki, jak światło UV, czy mitomicyna C, albo podczas wyboru ścieżki liza/lizogenizacja – w zależności od tego co jest dla faga bardziej korzystne w chwili infekcji.
Wybór cyklu lizogenicznego
W przypadku cyklu lizogenicznego bakteriofag produkuje białko CI. Białka CII oraz CIII, także są syntetyzowane i odpowiadają za utrzymanie stanu lizogenii. Po cyrkularyzacji DNA dochodzi do integracji materiału genetycznego faga z chromosomem gospodarza na zasadzie rekombinacji miejscowo-specyficznej.
Rekombinacja ma miejsce w wyznaczonych miejscach a są to attP po stronie bakteriofaga i attB po stronie chromosomu bakteryjnego. Reakcja ta jest katalizowana przez fagowe białko Int (integrase) oraz bakteryjne IHF (integration host factor). Po zintegrowaniu materiału genetycznego bakteriofaga i bakterii mówimy o profagu, który pozostaje stabilny i replikuje się wraz z genomem gospodarza.
Taka forma jednak nie musi pozostać na zawsze. Bakteriofag w odpowiednich warunkach może zostać wycięty z chromosomu bakteryjnego, mówimy wtedy o zjawisku indukcji. Zostaje uwolniony represor hamujący syntezę białka CI, co powoduje, że zachodzi ekspresja genów odpowiedzialnych za cykl lityczny. Wycięcie genomu fagowego jest prowadzone pod kontrolą kilku białek, między innymi: Int, Xis (kodowane przez faga) i Fis (kodowane przez gospodarza i odpowiadające za wiązanie DNA). Po wycięciu materiał faga ulega cyrkularyzacji i rozpoczynają się dalsze etapy cyklu litycznego.
Wybór cyklu litycznego
Na samym początku rozwoju cyklu litycznego DNA faga przeważnie replikuje się według modelu krąg po kręgu (ϴ), który później przekształca się w model toczącego się koła. W ten sposób tworzy się kilka cząsteczek DNA, co odpowiada kliku genomom DNA faga. Jednocześnie zachodzi generalna rekombinacja DNA fagowego i ekspresja genów faga (dokładnie kontrolowana przez różne mechanizmy), co prowadzi do produkcji białek kapsydu. Następnie dochodzi do pakowania, w którym konkatemeryczne cząsteczki DNA (czyli długi ciągły fragment DNA, który zawiera wiele kopii tej samej sekwencji DNA połączony szeregowo) są cięte do monomerów. Dojrzewanie kapsydów prowadzi do uformowania wirionów potomnych. Potomstwo fagowe uwalnia się z gospodarza po lizie, która spowodowana jest przez produkty genów faga.
Wykorzystanie bakteriofaga lambda
Badania przeprowadzone na bakteriofagu lambda oraz fagach lambdoidalnych doprowadziły do:
- poznania struktury genu i operonu oraz ich funkcji
- poznania mechanizmów kontroli ekspresji genów przez białka regulatorowe i małe RNA
- poznania białek opiekuńczych
- poznania mechanizmu wyboru ścieżki liza/lizogenia i ich poszczególnych etapów
- zaprojektowania techniki klonowania molekularnego
Nie są to wszystkie aspekty, które dotąd odkryto i z pewnością wciąż może ich przybyć. Jest to doskonały przykład tego, jak wiele można się dowiedzieć pracując na jednym modelu badawczym.
Lieratura i linki źródłowe
- Casjens SR, Hendrix RW. Bacteriophage lambda: Early pioneer and still relevant. Virology. 2015 May;479-480:310-30. doi: 10.1016/j.virol.2015.02.010. Epub 2015 Mar 3. PMID: 25742714; PMCID: PMC4424060.
- Catalano, Carlos E. „Bacteriophage lambda: The path from biology to theranostic agent.” Wiley Interdisciplinary Reviews: Nanomedicine and Nanobiotechnology 10.5 (2018): e1517.
- Hohn T, Katsura I. Structure and assembly of bacteriophage lambda. Curr Top Microbiol Immunol. 1977;78:69-110. doi: 10.1007/978-3-642-66800-5_3. PMID: 340151.
- Lederberg EM, Lederberg J. Genetic Studies of Lysogenicity in Escherichia Coli. Genetics. 1953 Jan;38(1):51-64. doi: 10.1093/genetics/38.1.51. PMID: 17247421; PMCID: PMC1209586.
- Kellenberger, Edward. „Vegetative bacteriophage and the maturation of the virus particles.” Advances in virus research. Vol. 8. Academic Press, 1962. 1-61.
- Kemp, C. L., A. F. Howatson, and L. Siminovitch. „Electron microscope studies of mutants of lambda bacteriophage: I. General description and quantitation of viral products.” Virology 36.3 (1968): 490-502.
- Morse ML, Lederberg EM, Lederberg J. Transduction in Escherichia Coli K-12. Genetics. 1956 Jan;41(1):142-56. doi: 10.1093/genetics/41.1.142. PMID: 17247607; PMCID: PMC1209761.
- Seah NE, Warren D, Tong W, Laxmikanthan G, Van Duyne GD, Landy A. Nucleoprotein architectures regulating the directionality of viral integration and excision. Proc Natl Acad Sci U S A. 2014 Aug 26;111(34):12372-7. doi: 10.1073/pnas.1413019111. Epub 2014 Aug 11. PMID: 25114241; PMCID: PMC4151757.
- Tal A, Arbel-Goren R, Costantino N, Court DL, Stavans J. Location of the unique integration site on an Escherichia coli chromosome by bacteriophage lambda DNA in vivo. Proc Natl Acad Sci U S A. 2014 May 20;111(20):7308-12. doi: 10.1073/pnas.1324066111. Epub 2014 May 5. PMID: 24799672; PMCID: PMC4034188.
- Walker PJ, Siddell SG, Lefkowitz EJ, Mushegian AR, Adriaenssens EM, Alfenas-Zerbini P, Davison AJ, Dempsey DM, Dutilh BE, García ML, Harrach B, Harrison RL, Hendrickson RC, Junglen S, Knowles NJ, Krupovic M, Kuhn JH, Lambert AJ, Łobocka M, Nibert ML, Oksanen HM, Orton RJ, Robertson DL, Rubino L, Sabanadzovic S, Simmonds P, Smith DB, Suzuki N, Van Dooerslaer K, Vandamme AM, Varsani A, Zerbini FM. Changes to virus taxonomy and to the International Code of Virus Classification and Nomenclature ratified by the International Committee on Taxonomy of Viruses (2021). Arch Virol. 2021 Sep;166(9):2633-2648. DOI: 10.1007/s00705-021-05156-1.
- Weigle, Jean J., and Max Delbrück. „Mutual exclusion between an infecting phage and a carried phage.” Journal of bacteriology 62.3 (1951): 301-318.
- Węgrzyn G, Licznerska K, Węgrzyn A. Phage λ–new insights into regulatory circuits. Adv Virus Res. 2012;82:155-78. doi: 10.1016/B978-0-12-394621-8.00016-9. PMID: 22420854.
- Wurtz M, Kistler J, Hohn T. Surface structure of in vitro assembled bacteriophage lambda polyheads. J Mol Biol. 1976 Feb 15;101(1):39-56. doi: 10.1016/0022-2836(76)90065-6. PMID: 1255721.
- https://ictv.global/taxonomy/taxondetails?taxnode_id=202101072