Czym jest wielolekooporność bakterii i jakie są dla nas najbardziej niebezpieczne? Czym jest antybiotyk ostatniej szansy? Patogeny ESKAPE, na nich skupimy się w poniższym artykule.
Spis treści
Wielolekooproność
Zacznijmy sobie od wyjaśnienia tego pojęcia. Wielolekooporność (ang. multidrug-resistant – MDR) bakterii to oporność na więcej niż 3 klasy antybiotyków. Na całym świecie jest to obecnie problem, z którym musimy sobie poradzić. W przypadku bakterii jest to normalne, że wytwarzają mechanizmy oporności, bo jest to ich sposób na przystosowanie się do życia w określonych warunkach i geny oporności występują
w środowisku naturalnie. W przypadku wykorzystania antybiotyków doprowadziliśmy do pewnej selekcji genów oporności anty-mikrobiologicznej. Dla nas jednak to znaczy, że musimy szukać nowych rozwiązań do walki z groźnymi patogenami [1].
ESKAPE
Światowa Organizacja Zdrowia (ang. World Helath Organization – WHO) wylistowała patogeny, dla których pilnie potrzebujemy nowych metod do ich zwalczania. W skrócie nazywamy je ESKAPE, od pierwszych liter nazwy rodzajowej bakterii.
E – Enterococcus faecium
S – Staphylococcus aureus
K – Klebsiella pneumoniae
A – Acinetobacter baumannii
P – Pseudomonas aeruginosa
E – Enterobacter species
Przez brak metod diagnostycznych, dzięki którym w szybki sposób moglibyśmy identyfikować bakterie oraz geny AMR często niepotrzebnie używa się antybiotyków o szerokim spektrum. To tylko pogłębia problem, ponieważ coraz więcej bakterii może nabyć nowe cechy oporności[1, 2].
Szacuje się, że w Europie rocznie przez zakażenia szpitalne oraz nabyte przez społeczność infekcje AMR umiera 33 000 osób i 874 000 lat życia skorygowanych niepełnosprawnością (wskaźnik DALY ang. disability-adjusted life years, stosowany do określenia stanu zdrowia w danym społeczeństwie) też są przez te zakażenia wywołane. Powoduje to ogromne koszty leczenia, które szacuje się na około
1,5 miliarda dolarów rocznie.
Bakterie wykształciły oporność poprzez mutacje genetyczne a także ruchome elementy genetyczne (jak plazmidy czy transpozony). Parogeny ESKAPE wykształciły mechanizmy oporności (Ryc. 1) na wiele klas antybiotyków: oksazolidynony, lipopeptydy, makrolidy, fluorochinolony, tetracykliny, beta-laktamy, inhibitory Beta-laktamów-Beta-laktamaz a także na antybiotyki ostatniej szansy, m. in. karbapanemy, glikopeptydy oraz polimyksyny (np. kolistyna, ale te nie są klinicznie korzystne) [1, 3].
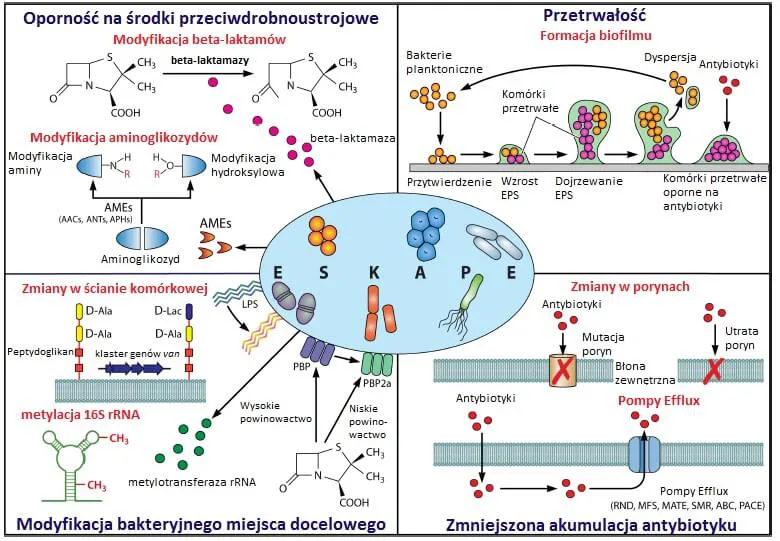
Antybiotyk ostatniej szansy
Bakterie, takie jak wymienione powyżej, mogą wykazywać oporność na wiele klas antybiotyków. Czasem zostaje ostatnie wyjście, czyli zastosowanie tzw. antybiotyku ostatniej szansy (mówi się też o nich antybiotyki ostatniej linii obrony). Używa się ich ostatecznie, abyśmy też nie nadużyli ich znów doprowadzając do oporności także na tę klasę antybiotyków.
Najczęściej mówi się tak o polimyksynach (np. polimyksyna B, kolistyna). Jest to klasa antybiotyków lipopeptydowych, które były stosowane już w późnych latach pięćdziesiątych XX w. W latach siedemdziesiątych ze względu na ich szkodliwość (nefrotoksyczność), a także dostępność już innych antybiotyków zrezygnowano z ich stosowania. Badania nad tymi antybiotykami zostały na długo porzucone, ale niedawno, ze względu na potrzebę ich zastosowania, zostały wznowione nowymi metodami między innymi w celu dobrania odpowiedniej dawki i jak najmniejszej szkodliwości dla pacjenta [4].
Opis bakterii ESKAPE
Pomimo tego, że wszystkie te bakterie są groźne dla naszego społeczeństwa ze względu na oporność na wiele leków, to wywołują one różne schorzenia. Scharakteryzujmy je sobie bardzo krótko.
Enterococcus
Bakterie z rodzaju Enterococcus to Gram-dodatnie ziarniaki (Ryc. 2) i fakultatywne anaeroby. Są one komensalami w naszym układzie pokarmowym (oznacza to między innymi, że naturalnie występuje
w naszym organizmie). Dla osób zdrowych te bakterie są nieszkodliwe (o takiej bakterii mówi się, że jest patogenem oportunistycznym). Inaczej jest u osób z obniżoną odpornością układu immunologicznego. Odpowiadają za zakażenia dróg moczowych związanych z cewnikiem, zapalenie wsierdzia, czy bakteriemię.
Stosowanie antybiotyków o szerokim spektrum doprowadziło do tego, że nasze jelita zostały skolonizowane przez E. faecium. To zwiększyło ilość bakterii Gram-ujemnych występujących naszej mikrobiocie. Bakterie te wykazują szczególnie często oporność na beta-laktamy. Wielolekooporne
E. faecium wykazują oporność także na aminoklikozydy (np. tobramycynę, kanamycynę, gentamycynę), streptomycynę, czy fluorochinolony [2].
Staphylococcus aureus
Jest to Gram-dodatni ziarniak (Ryc. 3), który jest fakultatywnym anaerobem. Powoduje różne infekcje, od łagodnych po te zagrażające życiu. So to między innymi infekcje skóry i tkanek miękkich, bakteryjne zapalenie wsierdzia, infekcje opłucnowe i infekcje związane z urządzeniami (na urządzeniach może powstać biofilm, który jest bardzo trudny do usunięcia i ze względu na swoją strukturę raczej niewrażliwy na działanie antybiotyków).
Pierwszą nabytą przez tę bakterię oporność odkryto bardzo szybko, bo już po 3 latach po odkryciu penicyliny – bakterie posiadały plazmid kodujący beta-laktamazę. W roku 1961 zaobserwowano pierwsze bakterie S. aureus oporne na metycylinę (ang. Methycylin Resistnat Staphylococcus aureus – MRSA),
a ostatnio także pojawiły się szczepy oporne na wankomycynę (ang. Vancomycin Resistant Staphylococcus aureus – VRSA). Bakterie te wykazują także oporność na fluorochinolony, daptomycynę, erytromycynę i wiele innych antybiotyków [2].
Klebsiella pneumoniae
Jest to Gram-ujemna pałeczka posiadająca otoczkę i zdolna do wytwarzania biofilmu (Ryc. 4.). Jest powodem takich chorób jak: infekcje układu moczowego, zapalenie płuc, ropień wątroby, zakażenia pooperacyjne i zakażenia krwi szczególnie u osób z obniżoną odpornością.
Bakteria ta stała się oporna na antybiotyki przez nabycie genów kodujących enzymy, które je degradują. Są to głównie beta-laktamazy o rozszerzonym spektrum (ang. Extended-Spectrum Beta-Lactamase – ESBL). i karbapenemazy. Karbapenemy to też często antybiotyki ostatniej linii obrony na uporczywe infekcje powodowane przez Gram-ujemne bakterie. To właśnie ten szczep K. pneumoniae, który wytwarza kabapenemazę stanowi istotne zagrożenie dla zdrowia publicznego [2].
Acinetobacter baumannii
Jest to Gram-ujemna pałeczka (Ryc. 5.) tlenowa i oportunistyczny patogen (występuje naturalnie w naszej mikrobiocie, ale u osób z obniżoną opornością może doprowadzić do wywołania choroby). Głównie wywołuje zakażenia szpitalne, takie jak respiratorowe zapalenie płuc, infekcje dróg moczowych, zapalenie opon mózgowych, bakteriemię a także infekcje przewodu pokarmowego, infekcje skóry, albo ran [5].
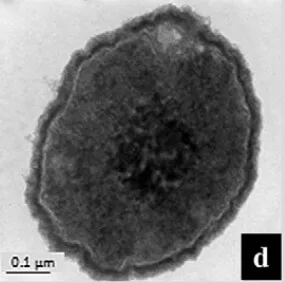
A. baumannii wykazuje wiele mechanizmów oporności, a są to: produkcja enzymów degradujących antybiotyki beta-laktamowe, działanie pomp efflux, enzymatyczna modyfikacja aminoglikozydów, produkcja zmodyfikowanych poryn, co zmniejsza przepuszczalność błony zewnętrznej oraz zmiana celu dla antybiotyku.
Bakterie te stały się też oporne na karbapenemy. W zależności od oporności danego szczepu stosuje się mieszanki różnych antybiotyków w celu ich zwalczenia [2].
Pseudomonas aeruginosa
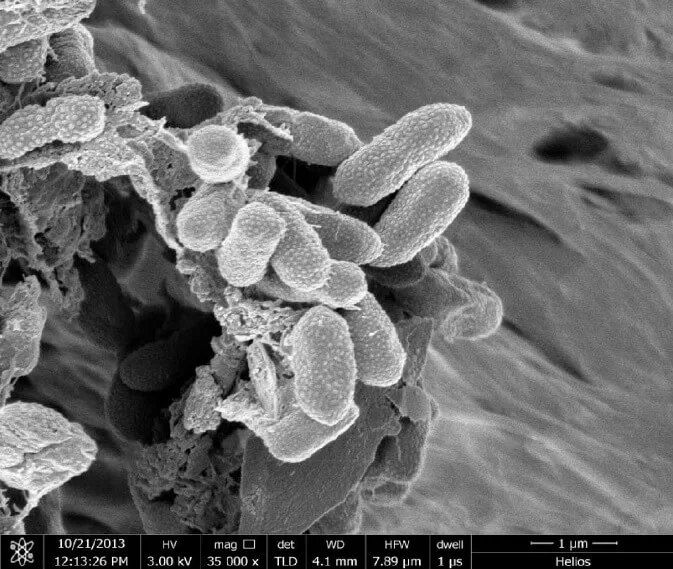
To także jest Gram-ujemna pałeczka (Ryc. 6) tlenowa (w naszym języku znana jako pałeczka ropy błękitnej). Występuje w środowisku i może odpowiadać za ostre i przewlekłe zakażenia szpitalne a w tym ciężkie infekcje układu oddechowego u pacjentów z upośledzoną odpornością oraz szpitalne zakażenia krwi [2]. Infekuje także trakt moczowy i jest najczęstszą przyczyną infekcji oparzeniowych, może też prowadzić do infekcji ucha zewnętrznego (co potocznie nazywamy uchem pływaka). Ma zdolność do tworzenia biofilmów, dlatego bardzo dobrze kolonizuje np. sprzęt medyczny. P. aeruginosa to także jeden
z najczęściej izolowanych patogenów z dróg oddechowych pacjentów z mukowiscydozą [6].
Główne mechanizmy oporności u tego patogenu, to wykorzystanie pomp efflux, obniżona przepuszczalność błony zewnętrznej a także nabycie, albo mutacja w genach kodujących białka, które kontrolują pasywną dyfuzję antybiotyków przez błonę zewnętrzną. Oporność na karbapenemy także jest coraz częściej spotykana ze względu na obecność karbapenemaz. W Stanach Zjednoczonych 10-30% izolatów P. aeruginosa jest opornych na te antybiotyki [2, 7].
Bakterie te są oporne na wiele antybiotyków, a w tym beta-laktamy. Do ich zwalczenia u pacjenta używa się kombinacji kilku różnych antybiotyków.
Enterobacter spp.
Te gatunki bakterii to z kolei Gram-ujemne pałeczki (Ryc. 7) i fakultatywne beztlenowce [8]. Najczęściej infekcje powodowane są przez E. cloacae i E. aerogenes. E. aerogenes ze względu na swoje wyższe genotypowe podobieństwo do bakterii z rodzaju Klebsiella został przeklasyfikowany na ten rodzaj [2].
Są to patogeny oportunistyczne i do infekcji może dojść zwykle u hospitalizowanych osób z obniżoną odpornością. Zwykle są obecne na oddziałach intensywnej terapii, a szczególnie u pacjentów, którzy są wentylowani mechanicznie [8].
Oporność bakterii z rodzaju Enterobacter zwykle wynika z obecności enzymów degradujących antybiotyki jak, ESBL i karbapenemazy. Są oporne na wiele klas antybiotyków i w przypadku K. aerogenes obserwuje się oporność na kolistynę – co czyni tę bakterię szczególnie niebezpieczną [2].
Jak widać mechanizmy oporności bakterii na leki są przeróżne, dlatego tak istotne jest szukanie alternatywnych dróg leczenia. Mamy naprawdę wiele możliwości, ale potrzeba nam czasu, czy środków finansowych na badania. Bakteriofagi, enzymy fagowe i bakteryjne, olejki eteryczne i wiele innych opcji jest do rozważenia. W kolejnych artykułach opiszemy sobie poszczególne mechanizmy oporności występujące u bakterii a także różne metody zwalczania bakterii.
Literatura
- De Oliveira, D. M., Forde, B. M., Kidd, T. J., Harris, P. N., Schembri, M. A., Beatson, S. A., … & Walker, M. J. (2020). Antimicrobial resistance in ESKAPE pathogens. Clinical microbiology reviews, 33(3), e00181-19. DOI: 10.1128/CMR.00181-19.
- Mancuso, G., Midiri, A., Gerace, E., & Biondo, C. (2021). Bacterial antibiotic resistance: The most critical pathogens. Pathogens, 10(10), 1310. DOI: 10.3390/pathogens10101310.
- Partridge, S. R., Kwong, S. M., Firth, N., & Jensen, S. O. (2018). Mobile genetic elements associated with antimicrobial resistance. Clinical microbiology reviews, 31(4), e00088-17. DOI: 10.1128/CMR.00088-17.
- Nang, S. C., Azad, M. A., Velkov, T., Zhou, Q. T., & Li, J. (2021). Rescuing the last-line polymyxins: achievements and challenges. Pharmacological reviews, 73(2), 679-728. DOI: 10.1124/pharmrev.120.000020.
- Kyriakidis, I., Vasileiou, E., Pana, Z. D., & Tragiannidis, A. (2021). Acinetobacter baumannii antibiotic resistance mechanisms. Pathogens, 10(3), 373. DOI: 10.3390/pathogens10030373.
- Mielko, K. A., Jabłoński, S. J., Milczewska, J., Sands, D., Łukaszewicz, M., & Młynarz, P. (2019). Metabolomic studies of Pseudomonas aeruginosa. World Journal of Microbiology and Biotechnology, 35, 1-11. DOI: 10.1007/s11274-019-2739-1.
- Tenover, F. C., Nicolau, D. P., & Gill, C. M. (2022). Carbapenemase-producing Pseudomonas aeruginosa–an emerging challenge. Emerging microbes & infections, 11(1), 811-814. DOI: 10.1080/22221751.2022.2048972.
- Davin-Regli, A., & Pagès, J. M. (2015). Enterobacter aerogenes and Enterobacter cloacae; versatile bacterial pathogens confronting antibiotic treatment. Frontiers in microbiology, 6, 392. DOI: 10.3389/fmicb.2015.00392.
- Kim, S., Lee, H., Lee, S., Yoon, Y., & Choi, K. H. (2015). Antimicrobial action of oleanolic acid on Listeria monocytogenes, Enterococcus faecium, and Enterococcus faecalis. PLoS One, 10(3), e0118800. DOI: 10.1371/journal.pone.0118800.
- Mohamed, S. H., Mohamed, M. S., Khalil, M. S., Mohamed, W. S., & Mabrouk, M. I. (2018). Antibiofilm activity of papain enzyme against pathogenic Klebsiella pneumoniae. Journal of Applied Pharmaceutical Science, 8(6), 163-168. DOI: 10.7324/JAPS.2018.8621.
- Espinal, P., Martí, S., & Vila, J. (2012). Effect of biofilm formation on the survival of Acinetobacter baumannii on dry surfaces. Journal of Hospital Infection, 80(1), 56-60. DOI:10.1016/j.jhin.2011.08.013.
- Ng, W. J., Hing, C. L., Loo, C. B., Hoh, E. K., Loke, I. L., & Ee, K. Y. (2023). Ginger-Enriched Honey Attenuates Antibiotic Resistant Pseudomonas aeruginosa Quorum Sensing Virulence Factors and Biofilm Formation. Antibiotics, 12(7), 1123. DOI: 10.3390/antibiotics12071123.
- Tang, W., Song, L., Li, D., Qiao, J., Zhao, T., & Zhao, H. (2014). Production, characterization, and flocculation mechanism of cation independent, pH tolerant, and thermally stable bioflocculant from Enterobacter sp. ETH-2. PloS one, 9(12), e114591. DOI:10.1371/journal.pone.0114591.
